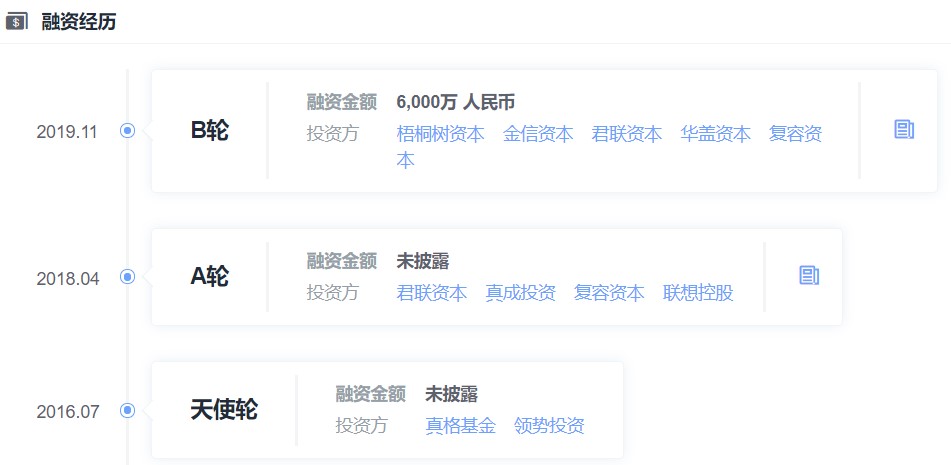
专注肿瘤病理诊断领域,「志诺维思」宣布获得六千万元 B 轮融资
据悉,「志诺维思」已完成六千万人民币B轮融资。此次轮次由华盖资本领投、梧桐树与清控金信蓝色微生物基金跟投;上一轮领投方君联资本、复容投资和公司高管团队在本轮均追加了投资。
「志诺维思」是一家 运用基因组信息和人工智能技术,为中国肿瘤精准医疗计划提供综合性信息解决方案和大数据平台的生物科技公司 。36氪了解到,「志诺维思」希望能够建立IVD检验机构建立肿瘤基因数据分析及解读的自动化体系、加速肿瘤临床和药研机构完成基于基因和病理数据的生物标记物发现,提升医院肿瘤病理的临床数据管理分析能力及科研水平。
截至目前,「志诺维思」产品及服务围绕精准医疗三大痛点进行布局:一、下一代肿瘤病理诊断革新(NGPD)的病理信息结构化;二、病理图像定量化;三、生信分析智能化。具体覆盖范围如下:
一、IT产品:个人基因组计算机——骇客TM(iGenomeComputerTM HiX)可以在15分钟内完成全基因组分析;精准医疗组学数据云平台——志诺云TM(iGenomeCloudTM)可以为医院、检测机构等提供基因组数据整理、分析、解读、传输等综合性解决方案;iCanSeqTM则是一套基因数据自动化分析和病历信息的智能化软件系统。
二、肿瘤异质性的临床检测产品AdamTar TM可应用于肿瘤临床治疗各个阶段。
三、抗癌登月:连接一百多家三甲医院、医生、患者及相关研究人员的肿瘤精准医学大数据平台。
36氪认为,「志诺维思」的竞争优势主要来源于:
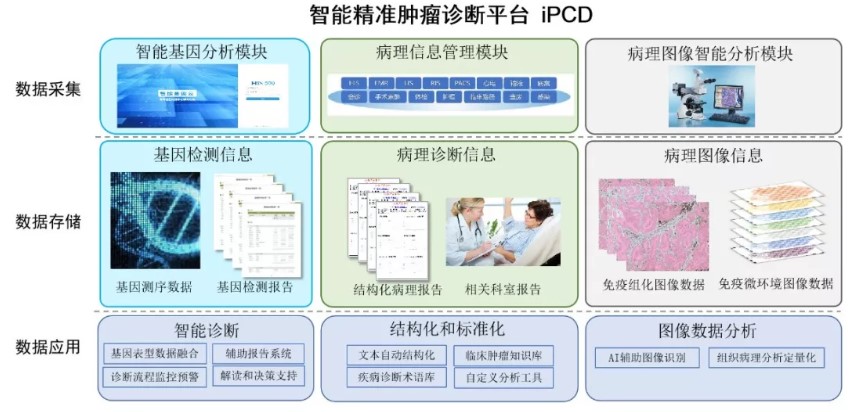
一、生信分析、自然语言处理和病理图像分析上的 自主知识产权的多模态AI技术体系。「志诺维思」的iPCD平台不仅包含之前智能基因云NGS的精准分析和临床解读功能,还可结合过往的病理信息帮助病理医生做出更精准的诊断,为临床医生呈现一份更具整合性的诊断和治疗建议报告。公开信息显示,「志诺维思」的技术在生产级大Panel数据和全外显子组(WES)上比现有开源算法高出15到20个百分点,数据分析的速率相较于开源算法可提高30到80倍,可满足临床应用的时效性和准确性要求。目前,头部基因测序设备厂商illumina和华大智造,都是「志诺维思」的合作伙伴。
二、平台效应。 「志诺维思」旗下的抗癌登月平台包含国际肿瘤基因组联盟(ICGC)的33个癌肿数万癌症患者的多组学数据及相应病理及临床信息,可为临床和药物研究提供参考依据。鉴于肿瘤病理诊断标准化是实现智能化和精准化诊断的第一步,通过这个平台携手全国病理专家共同实现多癌种的病理诊断标准化问题,「志诺维思」接下来将完成至少10个癌肿的标准化工作,为其未来发展打好基础。
鲸准数据显示,2016年至今,「志诺维思」共计完成三轮融资,总额过亿元。
目前,公司正积极推动下一代肿瘤病理诊断(Next-Gen Pathological Diagnosis:NGPD)革新,面向医疗机构打造全新的智能精准肿瘤诊断大数据平台(Intelligent Precision Cancer Diagnosis:iPCD)。本轮融资便是用于加快「志诺维思」iPCD平台的临床应用落地的步伐。
延伸阅读
人类治疗肿瘤的手段一直以来都在不断提升,药物治疗方式从早期的化疗、靶向小分子药物治疗到现在免疫检测点治疗、CART 治疗,精准医疗的治疗理念近十年得到了快速发展。
众所周知,病理诊断是肿瘤疾病诊断的金标准。随着精准医学进入肿瘤医学,其中精准医疗在病理诊断的应用(如分子诊断用于病理诊断的分子分型)能够更好地服务于病理诊断。
然而,由于病理诊断目前主要是手工操作主导,病理医生数量短缺成为限制病理行业发展的重要因素。行业普遍认为,AI 病理技术的出现有望解决这个问题。这是因为深度学习支撑的人工智能能够以迅速、标准化的方式处理医学影像,分辨出单个小区域内被标注为“肿瘤” 的像素,对可疑影像进行勾画、渲染,并以结构化的语音提供建议,辅助医生做出快速、精确的病理诊断。
在AI赋能病理领域方面,36氪了解到的玩家有「宜远智能」、「Reveal Biosciences」及「PathAI」等。
编辑:梦想家菜菜
图片来源:Pixabay
作者暂无likerid, 赞赏暂由本网站代持,当作者有likerid后会全部转账给作者(我们会尽力而为)。Tips: Until now, everytime you want to store your article, we will help you store it in Filecoin network. In the future, you can store it in Filecoin network using your own filecoin.
Support author:
Author's Filecoin address:
Or you can use Likecoin to support author: